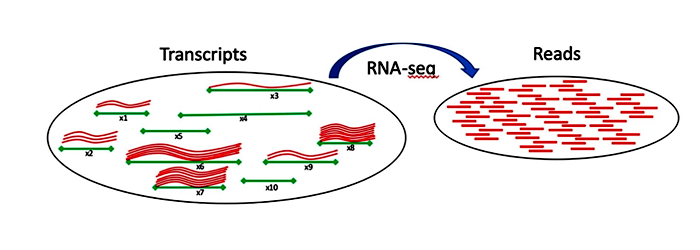
近日,中国农业科学院深圳农业基因组研究所农业宏基因组学创新团队联合国内科研单位开创性地提出了一种快速且全局最优的转录组定量解决方案TQSLE。该方法可准确定量序列高度相似的转录本的表达水平。相关研究成果发表在《生物信息学简报(Briefings in Bioinformatics)》上。
由于存在着序列极其相似的不同参考转录本,一部分转录组测序读段存在映射不确定性,从而无法通过映射读段的计数来估计转录本表达量。为解决这一问题,研究人员开发了一种非基于最大期望算法且非序列比对的方法TQSLE,为估计转录本和基因表达定量提供了一个全局最优的解决方案。在模拟及真实的转录组测序数据集上进行了一系列测试,结果表明,该技术相比基于最大期望算法的非序列比对的转录组定量方法更为准确。
该研究得到深圳市基础研究专项、国家自然科学基金等项目的资助。(通讯员 马昕怡)
原文链接:https://doi.org/10.1093/bib/bbad298